今回は,scikit-learnなどの既成ライブラリに頼らずに線形回帰モデルを実装していこうと思います。また,本記事はpython実践講座シリーズの内容になります。その他の記事は,こちらの「Python入門講座/実践講座まとめ」をご覧ください。
読みたい場所へジャンプ!
理論
線形回帰モデルの理論は,こちらの記事をご覧ください。基本的に,本記事では数式を示しません。

データセット
今回用いるデータセットは,scikit-learnが提供しているものを利用します。scikit-learnは主に「回帰用」「分類用」の2種類のデータセットを提供していますが,今回はその中で「回帰用」に当たる「ボストンの住宅価格」に関するデータを利用していきます。
| レコード(行)数 | 506 |
|---|---|
| カラム(列)数 | 14 |
| 用途 | 回帰 |
| URL | scikit-learn公式サイト |
| CRIM | 人口 1 人当たりの犯罪発生数 |
|---|---|
| ZN | 25,000 平方フィート以上の住居区画の占める割合 |
| INDUS | 小売業以外の商業が占める面積の割合 |
| CHAS | チャールズ川によるダミー変数 (1: 川の周辺, 0: それ以外) |
| NOX | NOx の濃度 |
| RM | 住居の平均部屋数 |
| AGE | 1940 年より前に建てられた物件の割合 |
| DIS | 5 つのボストン市の雇用施設からの距離 (重み付け済) |
| RAD | 環状高速道路へのアクセスしやすさ |
| TAX | $10,000 ドルあたりの不動産税率の総計 |
| PTRATIO | 町毎の児童と教師の比率 |
| B | 町毎の黒人 (Bk) の比率を次の式で表したもの。 1000(Bk – 0.63)^2 |
| LSTAT | 給与の低い職業に従事する人口の割合 (%) |
コード
最初に,コードを部分的に解説していった後に,全体のコードをお伝えしていこうと思います。
必要なライブラリのインポート
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
import seaborn as sns
from pandas import DataFrame
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import mean_squared_error
from sklearn.metrics import r2_scoreまずは,必要なライブラリをインポートしましょう。scikit-learnなどの既成ライブラリに頼らずに線形回帰モデルを実装したいのですが,モデル評価指標の計算のために「mean_squared_error」と「r2_score」もインポートしています。
データフレームの作成
boston = datasets.load_boston()
df = pd.DataFrame(boston.data, columns=boston.feature_names)
df['PRICE'] = boston.target
df.head()説明変数間の相関を確認
sns.pairplot(df)
各説明変数の関係について視覚的に確認します。この中でも,特に強い正の相関を持っている「RM」を説明変数として1つ選び,目的変数「PRICE」に対して線形回帰を行っていきましょう。
RMとPRICEの確認
sns.pairplot(df[['RM', 'PRICE']])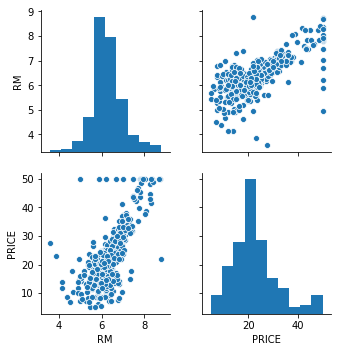
先ほどの全変数に対しての相関関係を見るのではなく,RMとPRICEにのみ注目したグラフを確認します。
基底関数の定義
def Polynomial(x, M):
phi_x = np.array([x**(j) for j in range(M+1)]).T
return phi_x
def Gaussian(X):
D = X.shape[0]
N = X.shape[1]
mu = np.array([[np.mean(X[j,:]) for j in range(D)]]).T
s = np.array([[np.sqrt(np.var(X[j, :])) for j in range(D)]]).T
phi_x = np.array(np.e**(-(X - mu)**2 / (2*s**2))).T
return [phi_x, mu, s]
def Sigmoid(X):
D = X.shape[0]
N = X.shape[1]
mu = np.array([[np.mean(X[j,:]) for j in range(D)]]).T
s = np.array([[np.sqrt(np.var(X[j, :])) for j in range(D)]]).T
a = (X - mu) / s
phi_x = 1/(1+np.e**(-a)).T
return [phi_x, mu, s]上から,多項式基底,ガウス基底,シグモイド基底になります。特に,多項式基底において$M=1$と設定すると,単項式回帰になります。すべての基底関数の出力は縦ベクトルで表し,ガウス基底とシグモイド基底の平均と標準偏差は,入力行列の「説明変数のサンプル数方向」に対して,それぞれ計算したものを利用します。ですので,平均と標準偏差は入力行列の「説明変数の種類」の数だけ生成されることになり,基底関数の数と一致します。
計画行列作成
def DesignMatrix(phi_X):
N = phi_X.shape[0]
phi_ones = np.ones(N).T
design_matrix = np.c_[phi_ones, phi_X]
return design_matrix計画行列を作成するための関数です。基底関数$\phi_0$は$1$と定めていますので,phi_onesというベクトルを,基底関数の出力の左側にくっつけています。
重みパラメータの最適化
def OptimalWeight(design_matrix, t):
optimal_weight = (np.linalg.inv(design_matrix.T @ design_matrix) @ (design_matrix.T)) @ t.T
return optimal_weight重みパラメータを最適化します。上でもとめた計画行列と,目的変数を引数として最適化された重みパラメータを返します。
モデルの出力
def Model(weight, phi_x):
y_out = weight.T @ phi_x
return y_out関数定義するまでもありませんが,入力に基底関数を施したもの(phi_x)と最適化された重みパラメータの内積が線形回帰モデルの出力となります。
データセットの分割
x_train, x_test, y_train, y_test = train_test_split(boston.data[:, [5]], boston.target, random_state=0)
x_train = x_train.T
x_test = x_test.T
y_train = y_train.T
y_test = y_test.T
x_train_poly = x_train.flatten()
x_test_poly = x_test.flatten()ボストンデータのRMに相当する列のみ取ってきます。そして,インポートしておいたsklearnのライブラリを利用して,ボストンデータを「75%の訓練(モデル学習用)データ」と「25%のテスト(モデル評価用)データ」に分割します。それぞれ,あとで都合がよいように転置を取って修正しています。また,多項式モデルでは入力が一次元として関数を定義していたので,そのように修正します。
モデルの学習
phi_X_poly1 = Polynomial(x_train_poly, 1)
phi_X_poly2 = Polynomial(x_train_poly, 2)
phi_X_poly3 = Polynomial(x_train_poly, 3)
phi_X_gauss = Gaussian(x_train)[0]
phi_X_sigm = Sigmoid(x_train)[0]
DM_poly1 = phi_X_poly1
DM_poly2 = phi_X_poly2
DM_poly3 = phi_X_poly3
DM_gauss = DesignMatrix(phi_X_gauss)
DM_sigm = DesignMatrix(phi_X_sigm)
w_opt_poly1 = OptimalWeight(DM_poly1, y_train)
w_opt_poly2 = OptimalWeight(DM_poly2, y_train)
w_opt_poly3 = OptimalWeight(DM_poly3, y_train)
w_opt_gauss = OptimalWeight(DM_gauss, y_train)
w_opt_sigm = OptimalWeight(DM_sigm, y_train)モデルを学習させます(=最適な重みパラメータを求めます)。今回は,多項式基底として$M=1,2,3$を利用しました。
モデルのテスト出力
phi_X_poly1_test = Polynomial(x_test_poly, 1)
phi_X_poly2_test = Polynomial(x_test_poly, 2)
phi_X_poly3_test = Polynomial(x_test_poly, 3)
phi_X_gauss_test = Gaussian(x_test)[0]
phi_X_sigm_test = Sigmoid(x_test)[0]
DM_poly1_test = phi_X_poly1_test
DM_poly2_test = phi_X_poly2_test
DM_poly3_test = phi_X_poly3_test
DM_gauss_test = DesignMatrix(phi_X_gauss_test)
DM_sigm_test = DesignMatrix(phi_X_sigm_test)
N_test = DM_poly1_test.shape[0]
y_poly1_test= np.array([Model(w_opt_poly1, DM_poly1_test[n,:]) for n in range(N_test)])
y_poly2_test= np.array([Model(w_opt_poly2, DM_poly2_test[n,:]) for n in range(N_test)])
y_poly3_test= np.array([Model(w_opt_poly3, DM_poly3_test[n,:]) for n in range(N_test)])
y_gauss_test = np.array([Model(w_opt_gauss, DM_gauss_test[n,:]) for n in range(N_test)])
y_sigm_test = np.array([Model(w_opt_sigm, DM_sigm_test[n,:]) for n in range(N_test)])訓練データを使って求めた最適な重みパラメータを利用して,テストデータから出力を求めます。
定量評価
mse_poly1 = mean_squared_error(y_poly1_test, y_test)
mse_poly2 = mean_squared_error(y_poly2_test, y_test)
mse_poly3 = mean_squared_error(y_poly3_test, y_test)
mse_gauss = mean_squared_error(y_gauss_test, y_test)
mse_sigm = mean_squared_error(y_sigm_test, y_test)
R2_poly1 = r2_score(y_poly1_test, y_test)
R2_poly2 = r2_score(y_poly2_test, y_test)
R2_poly3 = r2_score(y_poly3_test, y_test)
R2_gauss = r2_score(y_gauss_test, y_test)
R2_sigm = r2_score(y_sigm_test, y_test)
print('MSE_poly1 = ', mse_poly1)
print('MSE_poly2 = ', mse_poly2)
print('MSE_poly3 = ', mse_poly3)
print('MSE_gauss = ', mse_gauss)
print('MSE_sigm = ', mse_sigm)
print('R2_poly1 = ', R2_poly1)
print('R2_poly2 = ', R2_poly2)
print('R2_poly3 = ', R2_poly3)
print('R2_gauss = ', R2_gauss)
print('R2_sigm = ', R2_sigm)(以下は出力)
MSE_poly1 = 43.472041677202384
MSE_poly2 = 37.036872974321206
MSE_poly3 = 37.10046358044306
MSE_gauss = 72.13041721148669
MSE_sigm = 44.57442711949657
R2_poly1 = 0.04098622450766021
R2_poly2 = 0.27465819833584015
R2_poly3 = 0.22108417575897132
R2_gauss = -3.7688238397468767
R2_sigm = -0.1897487972972376求めたテストデータからの最適な出力と(推定結果),テストデータの目的変数(元から持っているデータ)を照らし合わせて,定量的な指標を計算します。今回は,平均二乗誤差(MSE)と決定係数(R^2)を利用しました。
既成のライブラリによる評価
from sklearn import linear_model
clf = linear_model.LinearRegression()
clf.fit(x_train.T, y_train.T)
pred = clf.predict(x_test.T)
mse = mean_squared_error(y_test.flatten(), pred)
R2 = clf.score(x_test.T, y_test.T)
print('mse = ', mse)
print('R^2 = ', R2)(以下は出力)
mse = 43.472041677202206
R^2 = 0.46790005431367815sklearnに入っている既成のライブラリを利用して線形回帰をしてみました。自作のモデルとの比較で利用できそうです。
視覚的な評価
xlist = np.arange(np.min(x_train), np.max(x_train), 0.01)
Xlist = np.array([xlist])
ylist_poly1 = [Model(w_opt_poly1, Polynomial(x, 1)) for x in xlist]
ylist_poly2 = [Model(w_opt_poly2, Polynomial(x, 2)) for x in xlist]
ylist_poly3 = [Model(w_opt_poly3, Polynomial(x, 3)) for x in xlist]
DM_gauss_graph = DesignMatrix(Gaussian(Xlist)[0])
DM_sigm_graph = DesignMatrix(Sigmoid(Xlist)[0])
N_graph = DM_gauss_graph.shape[0]
ylist_gauss = np.array([Model(w_opt_gauss, DM_gauss_graph[n,:]) for n in range(N_graph)])
ylist_sigm = np.array([Model(w_opt_sigm, DM_sigm_graph[n,:]) for n in range(N_graph)])グラフを描くための準備をしています。自分もハマってしまったのですが,ここでxlistとして生のボストンデータを利用するとカクカクな折れ線グラフが出力されます。それでは,モデルの妥当性を視覚的に確認することができないので,xlistとして「説明変数の最小値から最大値までを0.01の間隔でサンプリングしていった」リストを用意しました。間隔は適当に定めてOKですが,あまりにも大きいとカクカクしてしまうので注意が必要です。
plt.plot(xlist, ylist_poly1)
plt.plot(x_train_poly, y_train, 'o')
plt.show()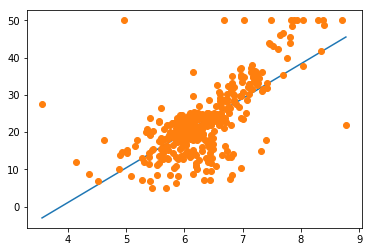
plt.plot(xlist, ylist_poly2)
plt.plot(x_train_poly, y_train, 'o')
plt.show()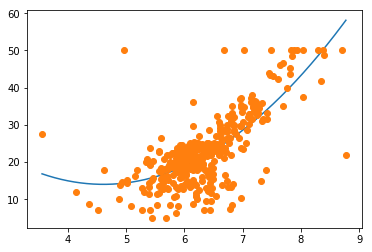
plt.plot(xlist, ylist_poly3)
plt.plot(x_train_poly, y_train, 'o')
plt.show()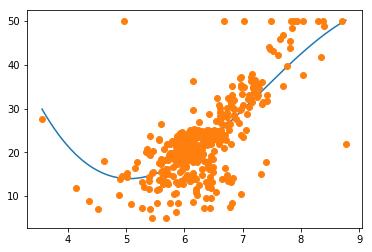
plt.plot(xlist, ylist_gauss)
plt.plot(x_train_poly, y_train, 'o')
plt.show()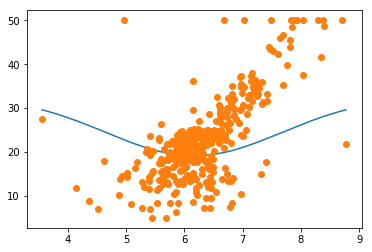
plt.plot(xlist, ylist_sigm)
plt.plot(x_train_poly, y_train, 'o')
plt.show()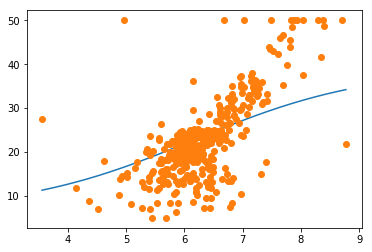
plt.plot(xlist, clf.predict(np.array([xlist]).T))
plt.plot(x_train_poly, y_train, 'o')
plt.show()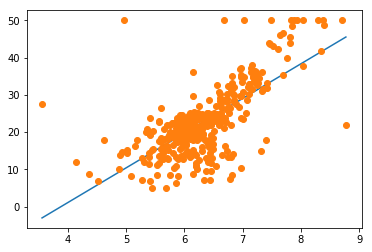
考察
今回は,ボストンデータの中から「RM(部屋数)」と「PRICE(価格)」の関係について注目しました。基底関数が多項式関数の場合,もちろん基底関数を増やすにしたがって滑らかなモデルが生成されますが,MSEとR^2を見てみると,多項式の次数が2の場合が最も定量的に良い結果が出ていることが分かります。
視覚的な確認により,多項式基底で$M=3$とした場合は,外れ値に敏感すぎる傾向にあることが分かります。明らかに,左側にポツンとある点に引きずられてしまっています。
また,既成のライブラリと比べて,今回のモデルの結果は芳しくありませんでした。理由としては,ガウス基底とシグモイド基底の個数が1つだったことが考えられます。今回の定義では,説明変数の個数とガウス基底とシグモイド基底の個数が一致するため,多くの説明変数を利用する重回帰分析になればなるほど,ガウス基底とシグモイド基底の威力が発揮されると思います。
ガウス基底とシグモイド基底で単回帰分析に対応するためには,基底の個数を固定してしまう(9個,など)という方法が考えられます。そうすることで,より妥当なモデルを構築することが可能になると考えられます。
もし理論にモヤモヤがあれば
(2026/02/22 04:57:54時点 楽天市場調べ-詳細)
こちらの参考書は,誰もが挫折しかけるPRMLよりも平易に機械学習全般の手法について解説しています。おすすめの1冊になりますので,ぜひお手に取って確認してみてください。
全コード
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
import seaborn as sns
from pandas import DataFrame
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import mean_squared_error
from sklearn.metrics import r2_score
boston = datasets.load_boston()
df = pd.DataFrame(boston.data, columns=boston.feature_names)
df['PRICE'] = boston.target
df.head()
sns_plot = sns.pairplot(df)
def Polynomial(x, M):
phi_x = np.array([x**(j) for j in range(M+1)]).T
return phi_x
def Gaussian(X):
D = X.shape[0]
N = X.shape
mu = np.array([[np.mean(X[j,:]) for j in range(D)]]).T
s = np.array([[np.sqrt(np.var(X[j, :])) for j in range(D)]]).T
phi_x = np.array(np.e**(-(X - mu)**2 / (2*s**2))).T
return [phi_x, mu, s]
def Sigmoid(X):
D = X.shape[0]
N = X.shape
mu = np.array([[np.mean(X[j,:]) for j in range(D)]]).T
s = np.array([[np.sqrt(np.var(X[j, :])) for j in range(D)]]).T
a = (X - mu) / s
phi_x = 1/(1+np.e**(-a)).T
return [phi_x, mu, s]
def DesignMatrix(phi_X):
N = phi_X.shape[0]
phi_ones = np.ones(N).T
design_matrix = np.c_[phi_ones, phi_X]
return design_matrix
def OptimalWeight(design_matrix, t):
optimal_weight = (np.linalg.inv(design_matrix.T @ design_matrix) @ (design_matrix.T)) @ t.T
return optimal_weight
def Model(weight, phi_x):
y_out = weight.T @ phi_x
return y_out
x_train, x_test, y_train, y_test = train_test_split(boston.data[:, [5]], boston.target, random_state=0)
x_train = x_train.T
x_test = x_test.T
y_train = y_train.T
y_test = y_test.T
x_train_poly = x_train.flatten()
x_test_poly = x_test.flatten()
phi_X_poly1 = Polynomial(x_train_poly, 1)
phi_X_poly2 = Polynomial(x_train_poly, 2)
phi_X_poly3 = Polynomial(x_train_poly, 3)
phi_X_gauss = Gaussian(x_train)[0]
phi_X_sigm = Sigmoid(x_train)[0]
DM_poly1 = phi_X_poly1
DM_poly2 = phi_X_poly2
DM_poly3 = phi_X_poly3
DM_gauss = DesignMatrix(phi_X_gauss)
DM_sigm = DesignMatrix(phi_X_sigm)
w_opt_poly1 = OptimalWeight(DM_poly1, y_train)
w_opt_poly2 = OptimalWeight(DM_poly2, y_train)
w_opt_poly3 = OptimalWeight(DM_poly3, y_train)
w_opt_gauss = OptimalWeight(DM_gauss, y_train)
w_opt_sigm = OptimalWeight(DM_sigm, y_train)
phi_X_poly1_test = Polynomial(x_test_poly, 1)
phi_X_poly2_test = Polynomial(x_test_poly, 2)
phi_X_poly3_test = Polynomial(x_test_poly, 3)
phi_X_gauss_test = Gaussian(x_test)[0]
phi_X_sigm_test = Sigmoid(x_test)[0]
DM_poly1_test = phi_X_poly1_test
DM_poly2_test = phi_X_poly2_test
DM_poly3_test = phi_X_poly3_test
DM_gauss_test = DesignMatrix(phi_X_gauss_test)
DM_sigm_test = DesignMatrix(phi_X_sigm_test)
N_test = DM_poly1_test.shape[0]
y_poly1_test= np.array([Model(w_opt_poly1, DM_poly1_test[n,:]) for n in range(N_test)])
y_poly2_test= np.array([Model(w_opt_poly2, DM_poly2_test[n,:]) for n in range(N_test)])
y_poly3_test= np.array([Model(w_opt_poly3, DM_poly3_test[n,:]) for n in range(N_test)])
y_gauss_test = np.array([Model(w_opt_gauss, DM_gauss_test[n,:]) for n in range(N_test)])
y_sigm_test = np.array([Model(w_opt_sigm, DM_sigm_test[n,:]) for n in range(N_test)])
mse_poly1 = mean_squared_error(y_poly1_test, y_test)
mse_poly2 = mean_squared_error(y_poly2_test, y_test)
mse_poly3 = mean_squared_error(y_poly3_test, y_test)
mse_gauss = mean_squared_error(y_gauss_test, y_test)
mse_sigm = mean_squared_error(y_sigm_test, y_test)
R2_poly1 = r2_score(y_poly1_test, y_test)
R2_poly2 = r2_score(y_poly2_test, y_test)
R2_poly3 = r2_score(y_poly3_test, y_test)
R2_gauss = r2_score(y_gauss_test, y_test)
R2_sigm = r2_score(y_sigm_test, y_test)
print('MSE_poly1 = ', mse_poly1)
print('MSE_poly2 = ', mse_poly2)
print('MSE_poly3 = ', mse_poly3)
print('MSE_gauss = ', mse_gauss)
print('MSE_sigm = ', mse_sigm)
print('R2_poly1 = ', R2_poly1)
print('R2_poly2 = ', R2_poly2)
print('R2_poly3 = ', R2_poly3)
print('R2_gauss = ', R2_gauss)
print('R2_sigm = ', R2_sigm)
from sklearn import linear_model
clf = linear_model.LinearRegression()
clf.fit(x_train.T, y_train.T)
pred = clf.predict(x_test.T)
mse = mean_squared_error(y_test.flatten(), pred)
R2 = clf.score(x_test.T, y_test.T)
print('mse = ', mse)
print('R^2 = ', R2)
xlist = np.arange(np.min(x_train), np.max(x_train), 0.01)
Xlist = np.array([xlist])
ylist_poly1 = [Model(w_opt_poly1, Polynomial(x, 1)) for x in xlist]
ylist_poly2 = [Model(w_opt_poly2, Polynomial(x, 2)) for x in xlist]
ylist_poly3 = [Model(w_opt_poly3, Polynomial(x, 3)) for x in xlist]
DM_gauss_graph = DesignMatrix(Gaussian(Xlist)[0])
DM_sigm_graph = DesignMatrix(Sigmoid(Xlist)[0])
N_graph = DM_gauss_graph.shape[0]
ylist_gauss = np.array([Model(w_opt_gauss, DM_gauss_graph[n,:]) for n in range(N_graph)])
ylist_sigm = np.array([Model(w_opt_sigm, DM_sigm_graph[n,:]) for n in range(N_graph)])
plt.plot(xlist, ylist_poly1)
plt.plot(x_train_poly, y_train, 'o')
plt.show()
plt.plot(xlist, ylist_poly2)
plt.plot(x_train_poly, y_train, 'o')
plt.show()
plt.plot(xlist, ylist_poly3)
plt.plot(x_train_poly, y_train, 'o')
plt.show()
plt.plot(xlist, ylist_gauss)
plt.plot(x_train_poly, y_train, 'o')
plt.show()
plt.plot(xlist, ylist_sigm)
plt.plot(x_train_poly, y_train, 'o')
plt.show()
plt.plot(xlist, clf.predict(np.array([xlist]).T))
plt.plot(x_train_poly, y_train, 'o')
plt.show()

















